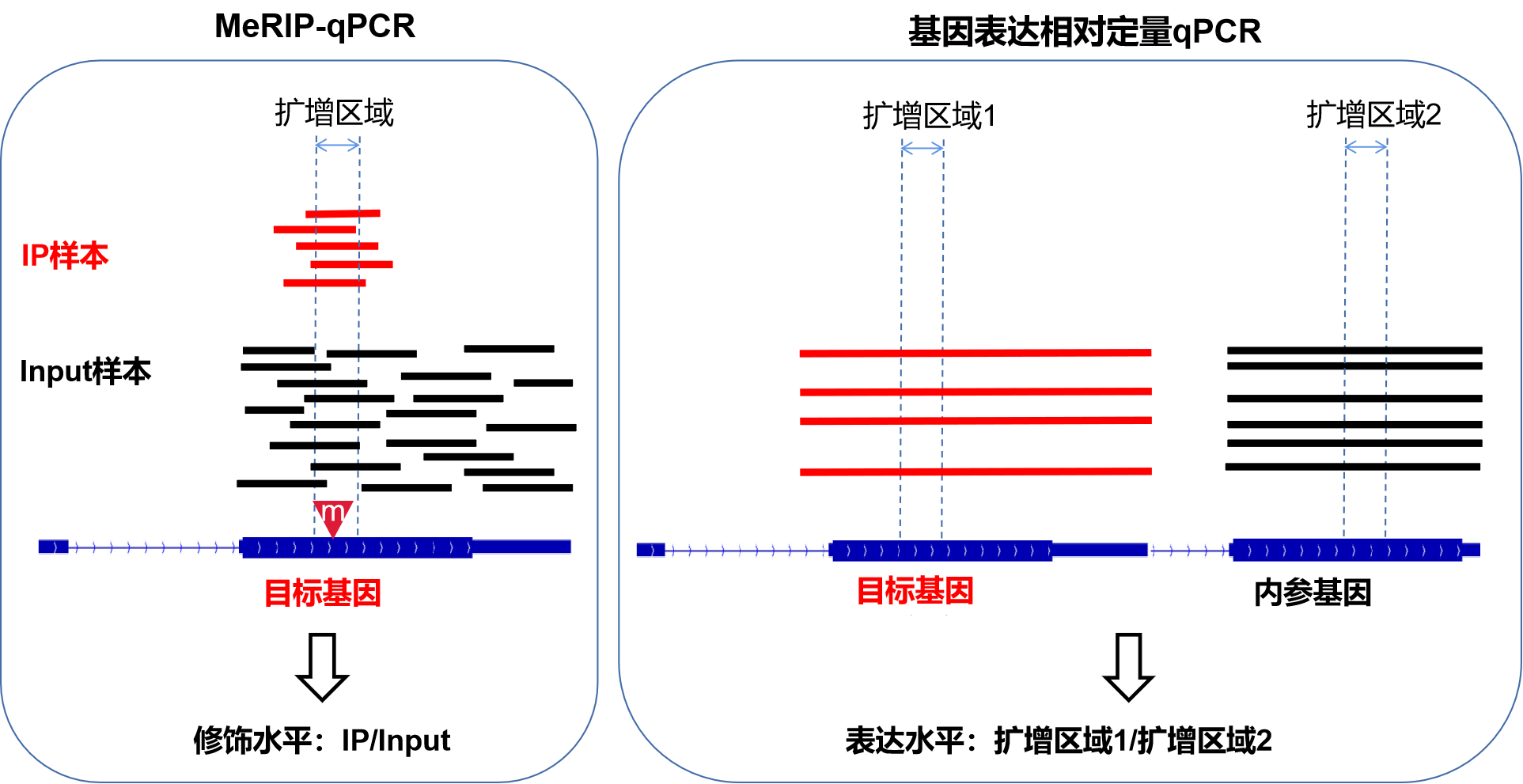
1.MeRIP-qPCR的目的是什么?
首先我们需要明确的是,MeRIP-qPCR的目的是验证某个目标基因上的某个修饰位点(以一小段区域的形式,MeRIP法精确不到单碱基)的修饰水平。
得到的结果往往是一个%(IP/Input)的数值,它体现的是该位点的修饰水平(可以大致理解为:这个转录本表达了很多条,其中百分之多少是这个位点有修饰的?)需要与普通qPCR验证基因表达水平(可以大致理解为:这个转录本表达了多少条?)的目的区分开。
2.MeRIP-qPCR的实验方法?
MeRIP-qPCR就是MeRIP实验后进行一个qPCR。
这里的MeRIP实验同MeRIP-seq测序中的其实相同:对RNA进行片段化。然后每个样品一分为二,一部分片段用针对该修饰的特异性抗体与RN**段进行免疫共沉淀(IP),留为IP样品,另一部分不进行IP直接留作Input样品。之后对两部分RN**段分别进行逆转录和qPCR。如果是测序,就是在这里把紧接的qPCR改为接建库测序。
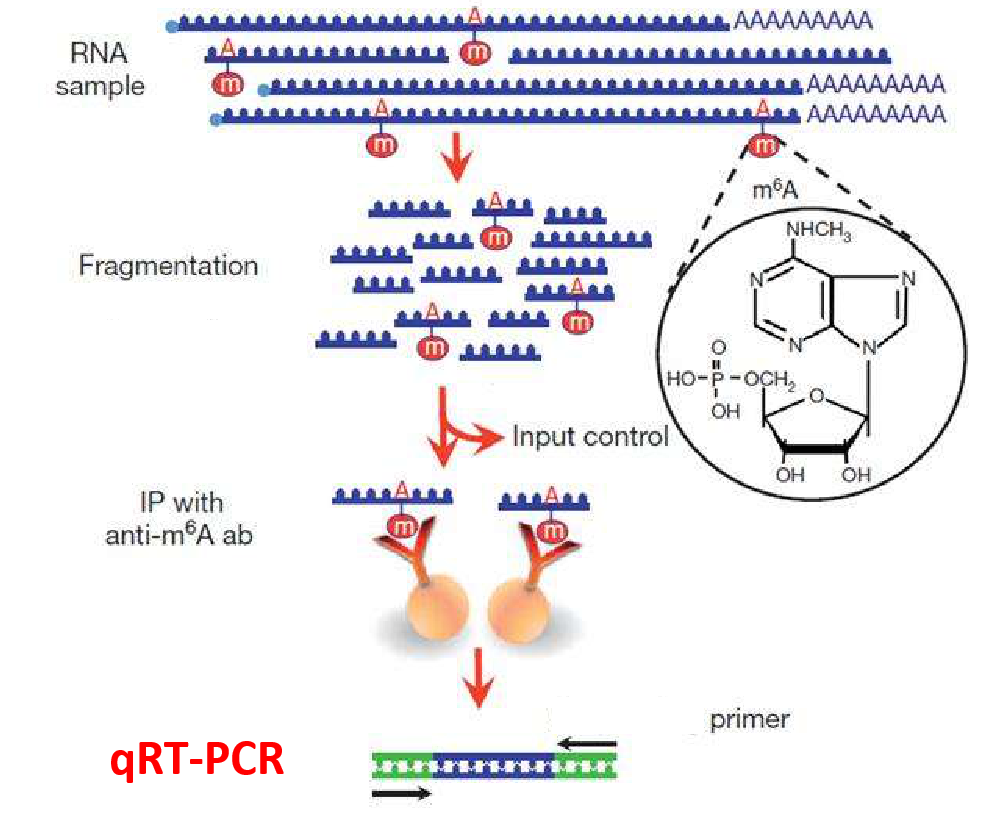
3.MeRIP-qPCR引物如何设计?
由于目的是定量位点的修饰水平,因此MeRIP-qPCR的引物不仅是针对目标基因,还要是针对其上的那个修饰位点(区域)。这个位点信息可以从:
(1)MeRIP-seq的测序结果中筛选得来
通常是一段位置坐标,比如云序生物提供的MeRIP-seq结果,某个甲基化峰位置如:chr1:111234-111567。有了这个位置信息,可以去UCSC genome browser网站:
http://genome.ucsc.edu/cgi-bin/hgGateway选对应的基因组版本获取这段序列。
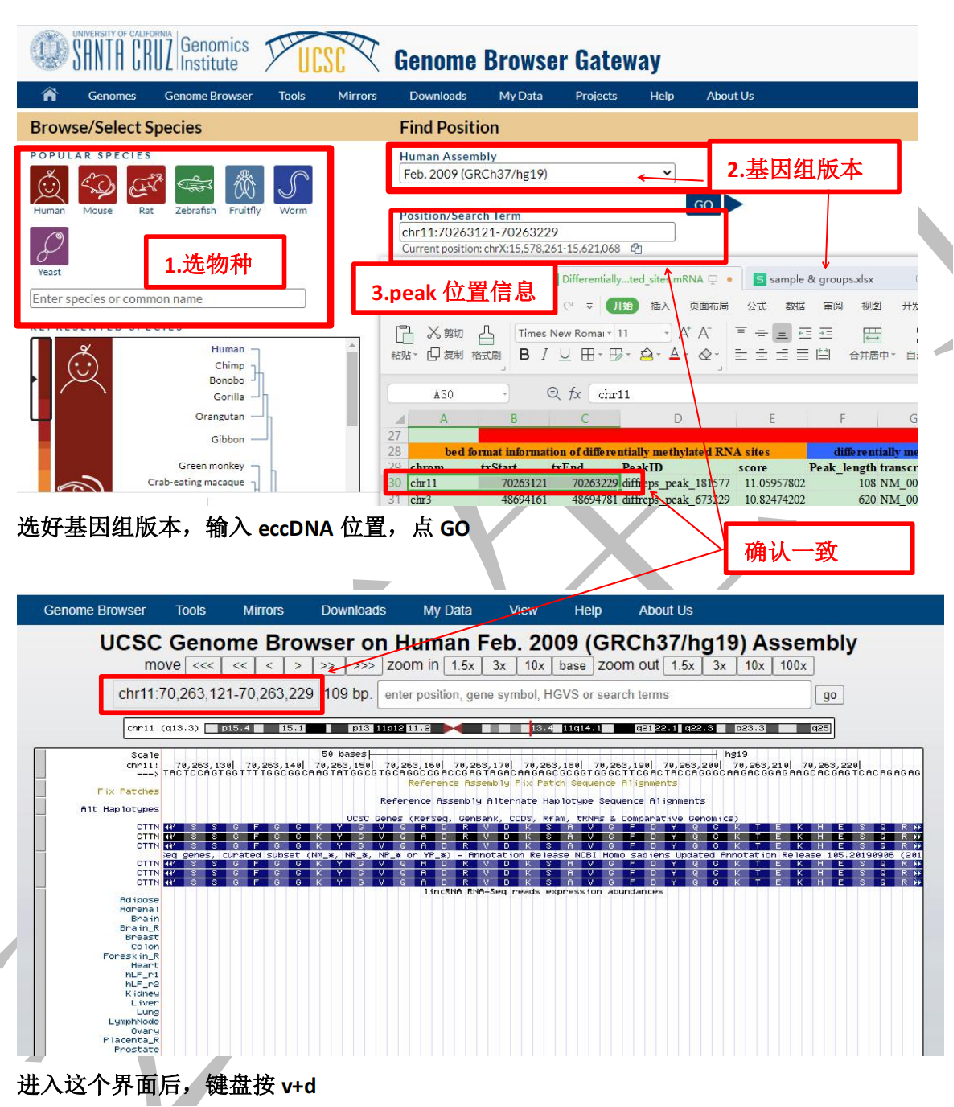

(2)网站预测得到
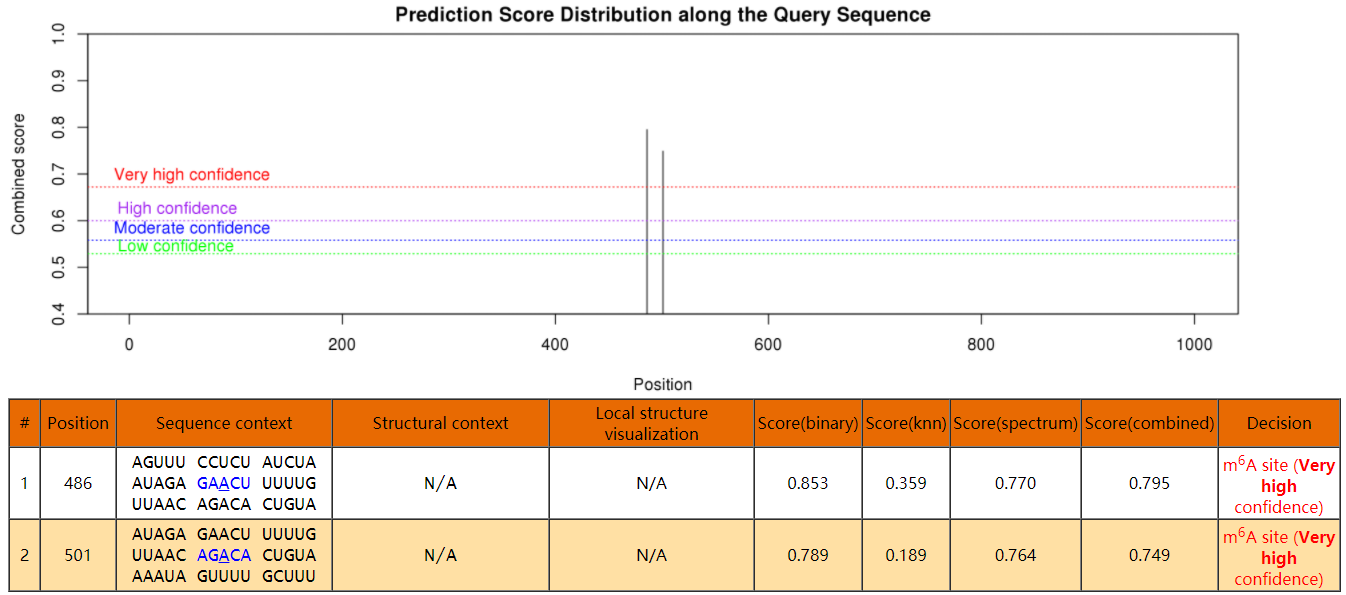
如果没有测序,想先通过MeRIP-qPCR看看关注的某基因上是否可能存在修饰,就只能预测一下发生修饰概率高的区域位置。通常这些网站是基于研究公认的该种修饰motif序列等进行预测,需要将目标RNA的序列输入,可生成预测结果。
m6A RNA甲基化预测网站:
http://www.cuilab.cn/sramp/
m5C RNA甲基化预测网站:
4.MeRIP-qPCR为什么没有内参基因?
(1)MeRIP-qPCR不需要内参来进行样本间的均一化矫正
从结果的形式%(IP/Input)我们也可以看到,这个实验侧重看修饰的片段(IP样品qPCR结果)占总片段(Input样品qPCR结果)的比例,实验目的只是要得到这两者的比值就体现修饰水平,因此MeRIP-qPCR不涉及到普通的相对定量qPCR需要的内参:
内参是衡量不同样品间表达量时用来均一化样品的,比如默认GAPDH在你的各个样本之间表达量应当相同,qPCR结果样品A中目的基因表达量1,GAPDH表达量10;样品B中目的基因表达量1.5,GAPDH表达量15,那么不能说因为1<1.5所以就说A样品目的基因表达量低,因为从GAPDH来看可能只是实验某环节中A样品的量整体少了一些而已。而是要看A中目的基因表达量是GAPDH的0.1倍,B也是0.1倍,两样品中目的基因表达量其实无差别。
换做MeRIP-qPCR中,如果针对目的基因目的位点qPCR,样品A的IP结果0.2,Input结果1;样品B的IP结果0.3,Input结果1.5,我们的%(IP/Input)结果显示两个样品甲基化水平数值都是0.2,修饰水平无差异,到此就完成了目的。
有人问那没有做内参,万一发生如上段提到的样品用量等环节有差异,会不会影响修饰水平的比较?显然,如果两个样品本来修饰水平相同,那么夸张地想,即使B样品用量由于手抖加了A的10倍,qPCR结果也会IP和Input信号都增长10倍,不影响这个比值即修饰水平。
前面第1点主要从多个样品比较的角度解释了MeRIP-qPCR为何不需要内参基因。
而有时出现以下情况:1)没有分组对比,只想看下这一个(组)样品中该基因算不算有修饰,此时有个疑问:结果的%(IP/Input)达到多少可算作有修饰?——回答是目前没有一个定值。那么此时难免会希望能有个已知有修饰的参考基因,我们的目的基因如果水平接近或者超过就算有修饰。2)想看看MeRIP实验体系有没有问题,做个已知有修饰的基因当阳性对照看看效果。然而RNA修饰是一个动态的可逆的酶促反应,在同个组织细胞中的水平可能都会随着环境千变万化,不同组织中更是差别很大,与普通qPCR检测基因表达时能找到稳定表达的看家基因不同,MeRIP-qPCR没有一个稳定修饰的基因做参照,目前RNA修饰文章也都不要求有这种参照。
针对上述目的1),可以同时多做一个非特异性IgG抗体的免疫沉淀富集,对比样品本身的IP和IgG是否有显著差异,放在文章中作为该位点有一定程度的修饰的证据。
针对目的2),也可做IgG对照检测体系的特异性。除此之外,云序生物代理的GenSeq m6A MeRIP试剂盒中除了提供了IgG抗体,还提供了一对m6A的阳参和一对阴参引物,是根据文献报道的,靶向 HEK293 细胞中 m6A 修饰的某段 RNA 区域(Positive Primers)和无 m6A 修饰的某段 RNA 区域(Negative Primers), 供有条件且感兴趣的用户验证实验效率和抗体特异性。但如前面所说,m6A 修饰具有很强的细胞类型特异性,无法保证在任何细胞系或样品中,该引物都能作为很好的参照。因此,建议想对实验体系进行进一步自验证的用户,采用 HEK293 细胞的 RNA 进行验证实验。


5.MeRIP-qPCR不能看出基因表达量?
有人想:Input样品又没经过IP,qPCR操作不就与普通qPCR没什么差别,那么做了MeRIP-qPCR,不能得到基因表达量的结果吗?比如前面MeRIP-qPCR例子中A和B样品Input分别是1和1.5的结果可以用来看一下这个基因在两样品中的表达量有没有差异?——我们不知道,因为没有GAPDH等内参基因来计算,要看表达量的话就不知道1和1.5的差异是否是如前一段那样由于用量差异等因素带来的了。因此通常说起MeRIP-qPCR,结果不能用来体现基因表达量。
那如果在MeRIP-qPCR步骤中直接多扩增一个GAPDH基因,与目的基因Input联合在一起定量表达量是否可行?我们只能说可以参考,不过由于MeRIP实验是有片段化这一步骤的,PCR信号会弱一些,对于验证表达量的目的来说还是推荐采用常规的qPCR步骤会更准确、风险更小。
6.IP/Input/IgG傻傻分不清:我需要做哪些、如何展示?
前面1.和2.两个环节谈到过,IP是用该修饰的特异性抗体,通过免疫共沉淀富集带修饰的片段,Input是不进行富集的所有片段,这两个是MeRIP-qPCR必做的,每个样品分别做了这两部分的PCR结果合起来计算才能体现修饰水平,达到实验的目的。而IgG如4(2)提到,是用非特异性抗体,通过免疫共沉淀富集非特异性结合的片段,作为对IP的对照,证明IP的信号不是源自抗体非特异性和,而是确实是针对发生修饰的片段。IgG可以选做。MeRIP-qPCR结果通常用柱状图展示即可,常见的如以下3种情况:
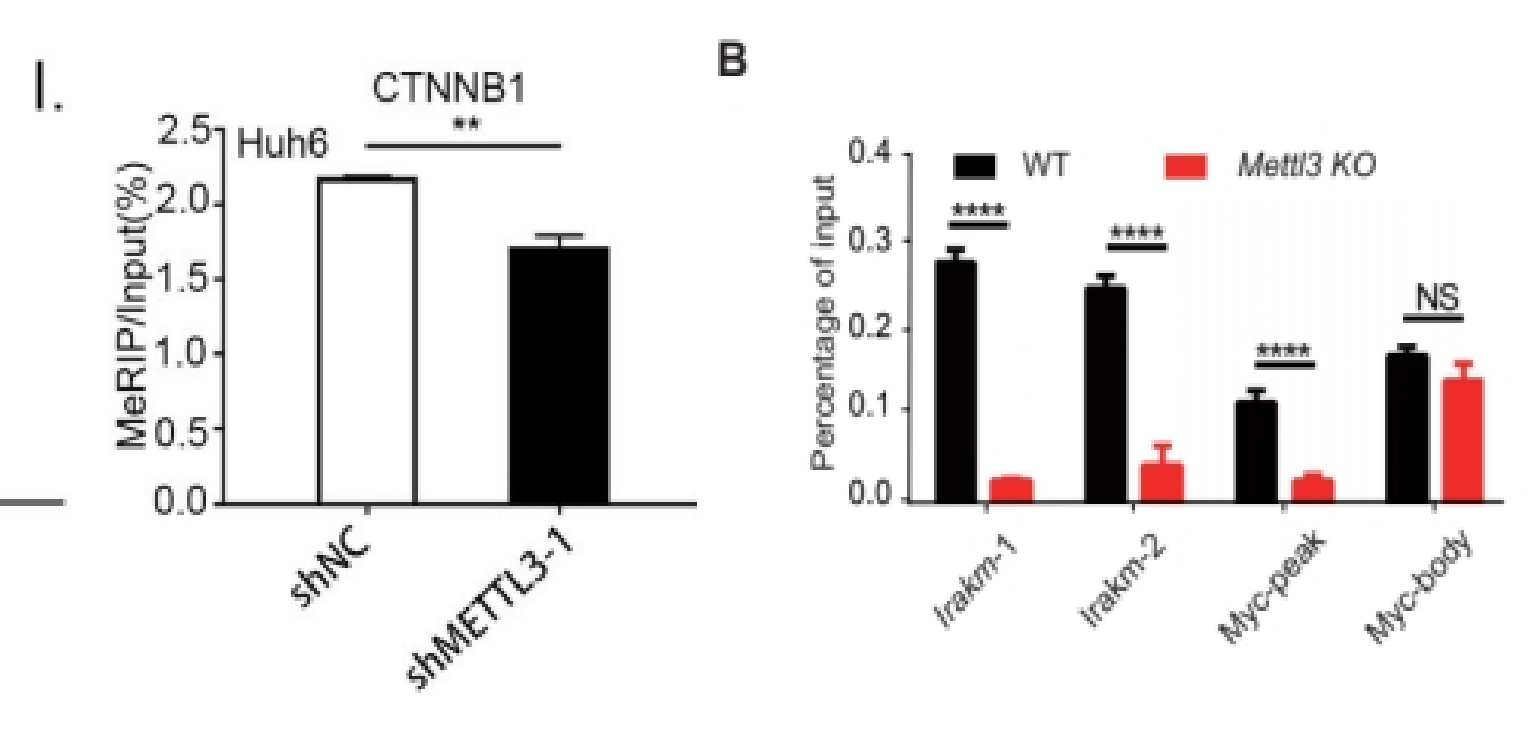
(1)只做IP和Input:展示%(IP/Input)值
如果有两组样品,展示一个目的基因在两组的修饰水平。或者一组样品,展示多个基因的修饰水平差异,可以只做IP和Input,展示%(IP/Input)值。[1][2]
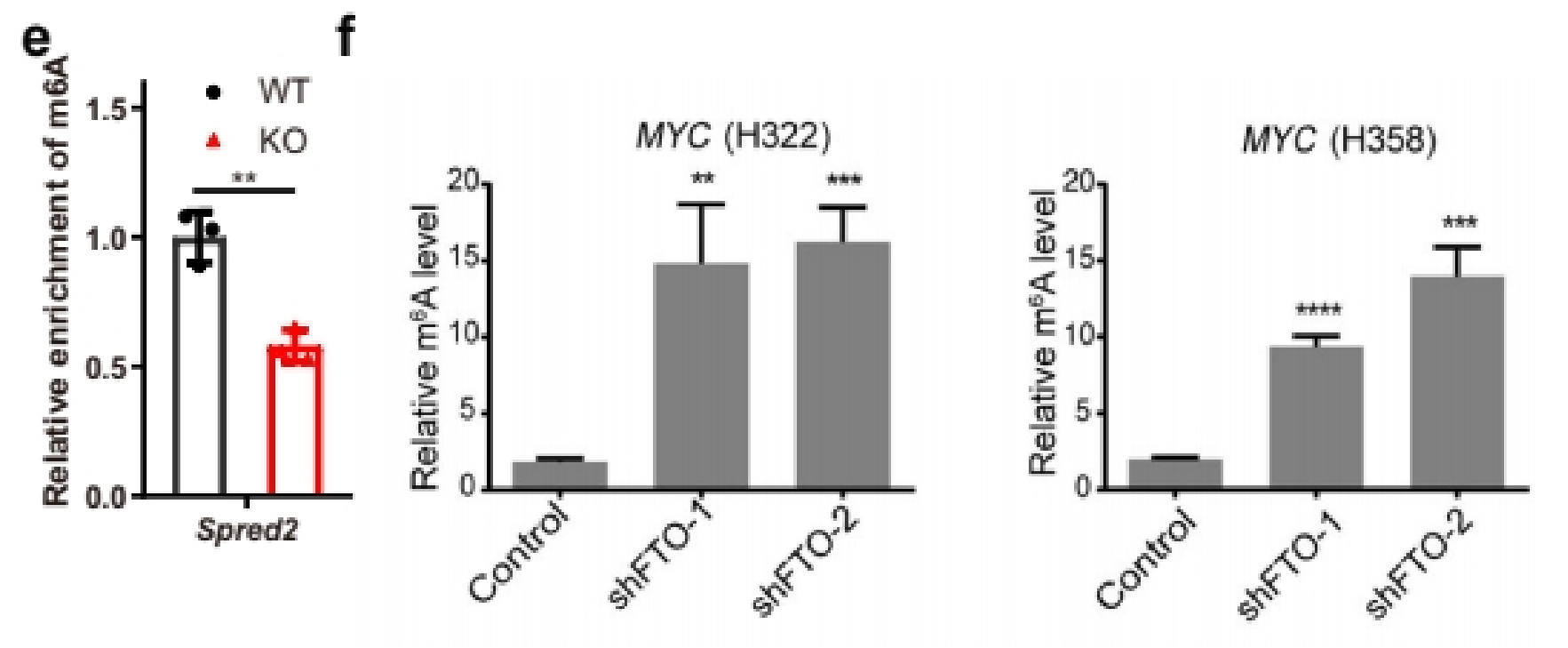
如果为了严谨的证明实验体系没有问题,或者验证指标只有一个分组/一个基因但需要一个对照,可以同时多做一个IgG,结果展示%(IP/Input)和%(IgG/Input)两个柱子对照。[5][6]
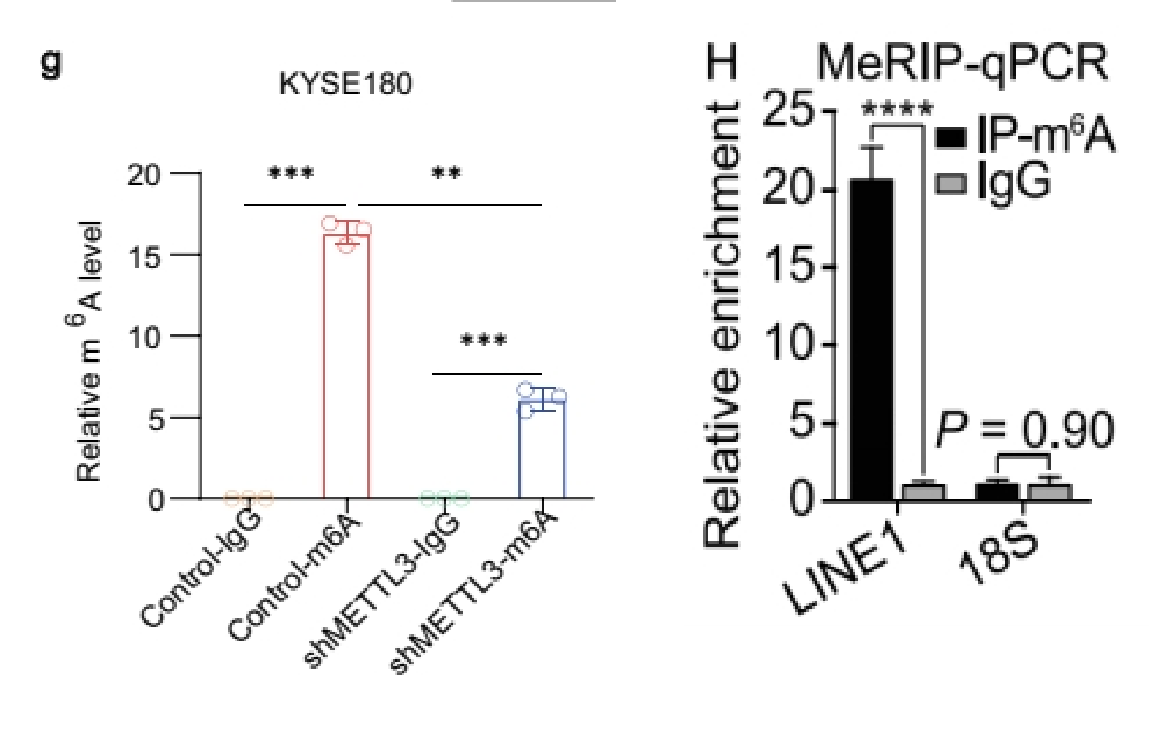
(3)做IP、IgG和Input:展示Fold enrichment值(%(IP/Input)和%(IgG/Input)的比值)
如果做了IgG对照,还可以选择用Fold Enrichment来展示,富集倍数%(IP/Input)/%( IgG/Input),即IP比对照的IgG信号高多少倍,也可以用来体现修饰的水平。通常是分组和基因都是多个时不想一一单独展示IgG。[7]
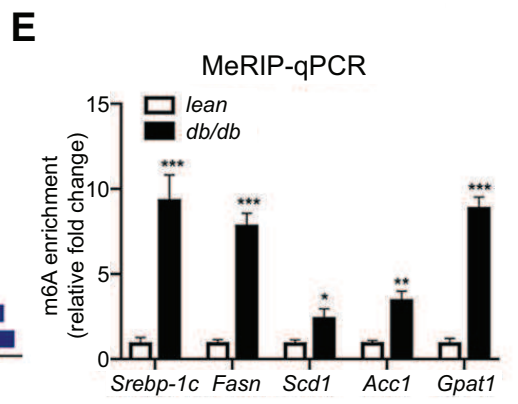
*以上图参考文献:
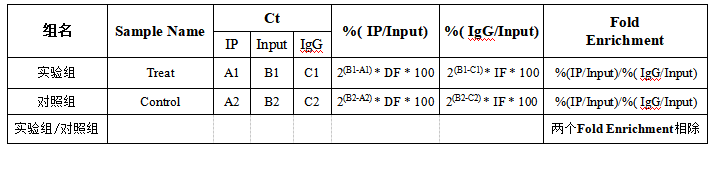
7.MeRIP-qPCR结果的计算?
MeRIP-qPCR结果计算如下表:
云序生物m6A修饰研究五大模块
01 m6ARNA修饰测序
m6A RNA修饰测序(m6A-meRIP-seq)
对m6A RNA甲基化,目前流行的检测手段为m6A-Seq技术,适用于m6A RNA甲基化谱研究,快速筛选m6A RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m6A测序:
● m6A 全转录组测序(涵盖mRNA,LncRNA,circRNA)
● m6A LncRNA测序(涵盖LncRNA和mRNA)
● m6A Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
● m6A mRNA测序
● m6A miRNA测序
02 检测整体m6ARNA修饰水平
LC-MS/MS检测整体RNA修饰水平
精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
比色法检测整体RNA修饰水平
快速检测m6A整体甲基化水平
03 m6A RNA修饰上游酶的筛选
m6A RNA修饰相关酶PCR芯片
寻找上游直接调控m6A RNA甲基化的甲基转移酶。
04 m6A RNA修饰靶基因验证
meRIP-qPCR
云序提供各类不同修饰的meRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
05机制互作研究
5.1 RIP-seq/qPCR
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
5.2 RNA pull down -MS/WB
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
5.3 双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
5.4 ChIP-seq
筛选或验证目标蛋白与DNA互作,研究相应的分子调控机制。
云序生物服务优势
优势一:发表10分以上文章最多的m6A RNA甲基化测序服务平台。云序已累计支持客户发表52+篇高水平文章,合计影响因子450分+,是国内支持发文最多、累计影响因子最高的公司。
优势二:至今完成4000+例 m6A测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:独家提供m6A一站式服务:m6A整体水平检测、m6A测序、MeRIP-qPCR验证、RIP和RNA pull-down等。
优势五:率先研发超微量MeRIP测序技术,RNA量低至500ng起。
优势六:国内最全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、O8G、ac4C乙酰化和2'-O-甲基化测序。
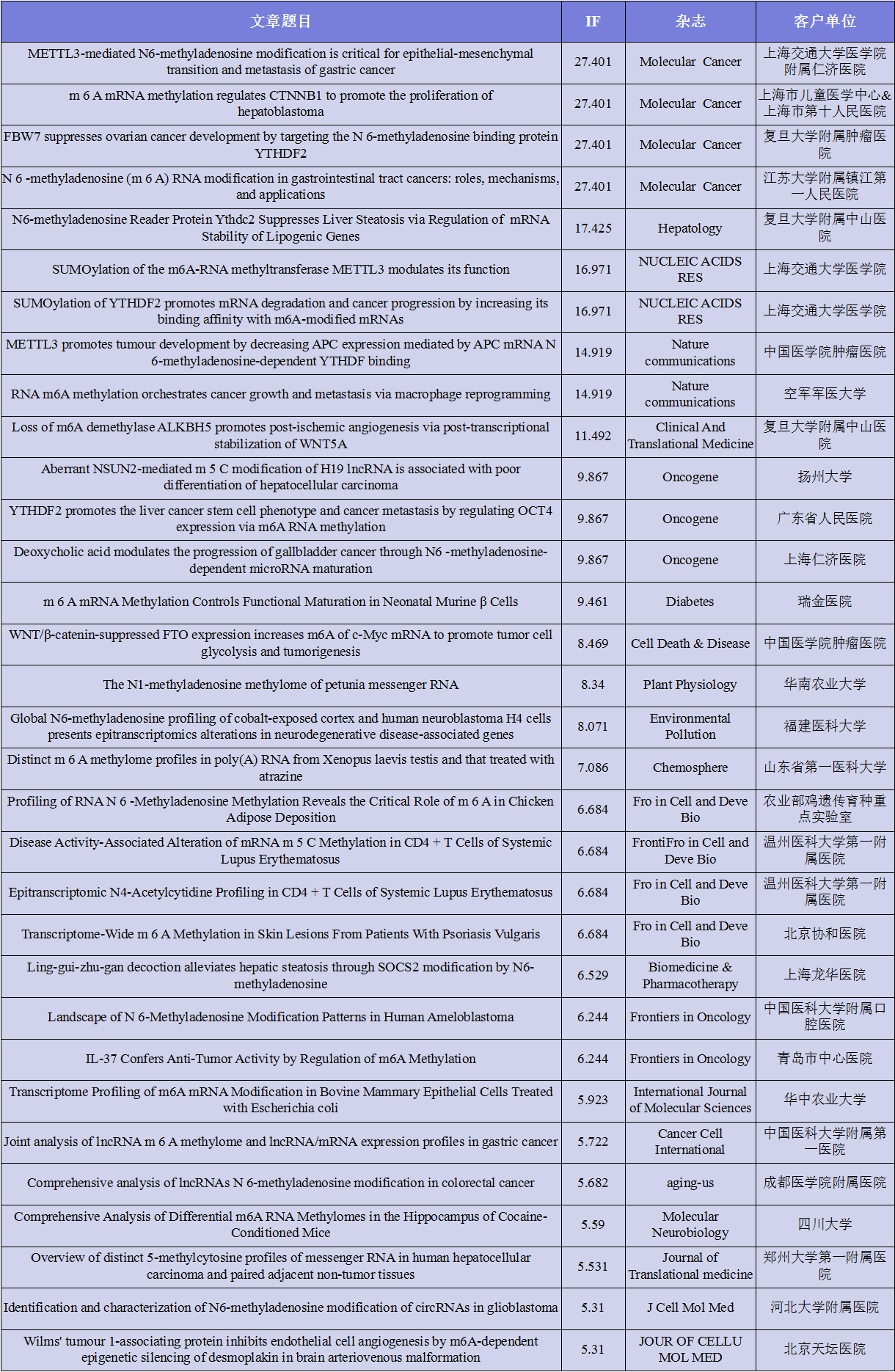
云序客户RNA修饰部分文章列表
相关产品
往期回顾
《Cell》新发现!m6A-MeRIP-seq技术揭示m6A 修饰新功能!
上海云序生物科技有限公司 商家主页
地 址: 上海市松江区莘砖公路518号24号楼4楼
联系人: 戴小姐
电 话: 021-64878766
传 真: 021-64878766
Email:market@cloud-seq.com.cn;liuqingqing@cloud-seq.com.cn
相关咨询
杨宝峰院士团队最新成果 | 云序助力揭示RNA修饰m7G调控心肌肥厚的机制研究 (2024-11-13T00:00 浏览数:2083)
Nature子刊| 重磅综述!一文总结「m6A修饰非编码RNAs」在各类肿瘤中的调控机制及作用 (暂无发布时间 浏览数:1855)
研究速览-eccDNA 2023年最新进展大放送! (暂无发布时间 浏览数:1683)
云序生物MeRIP-qPCR技术干货 (暂无发布时间 浏览数:1673)
技术干货| “eccDNA碱基序列的获取及引物设计”方法教程 (暂无发布时间 浏览数:1770)
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制 (暂无发布时间 浏览数:1637)
Nat Biotechnol IF=47 | BID-seq:一种基于单碱基分辨率的假尿嘧啶(Ψ)修饰定量测序检测方法 (暂无发布时间 浏览数:1293)
北大伊成器团队Nature Reviews重磅发文:非m6A热门修饰调控与功能一文速览! (暂无发布时间 浏览数:1835)
用户文章m6A专题|IF=9.8|m6A去甲基化酶ALKBH5缺乏会加重钴致神经退行性损伤 (暂无发布时间 浏览数:1139)
客户文章| Nature子刊 揭示了FMR1通过m6A修饰调控早期胚胎发育的分子机制 (暂无发布时间 浏览数:1742)
